7.雌ザル由来のVero細胞
私たちが行ったVero細胞の核型解析においては、その性染色体がXX型、すなわちメス型であることは一目瞭然です(図1)。ところが、Vero細胞が由来するサル個体の性別について言及している文献は、私たちの知る限り、一つも見当たりません。細胞樹立者である安村先生は、ワクチン製造所であった当時の千葉血清研究所から摘出腎臓を貰い受けたときに元の動物個体の性別は知らされず、Vero細胞の樹立後に他の研究者がおこなった核型解析でも性染色体セットを明確に決められずに今に至ってしまったのかもしれません(余談4)。
もともとはXY型(すなわちオス型)だったものが培養中にY染色体が脱落してXO型となり、このXが重複してXX型となったという可能性もありえましたが、この可能性は後で述べる全ゲノム配列決定により否定されました。X染色体重複が起こったとは考えられないほどの一塩基多様性(single nucleotide variations; SNVs)が、二本のX染色体間にはあるからです[1]。
8.ゲノム配列解読
さて、いよいよゲノム配列の解読です。と言っても実際には、核型解析を済ませてからゲノム配列の解読に進んだのではなく、これらは並行して進行しました。
高性能のシーケンサを用いれば、哺乳動物のゲノムであってもそのドラフト配列を決定するだけのシーケンス量を得ることは今や困難なことではありません。しかし、その際に必要となるDNAライブラリを作製することには多くのknow-howのいる手作業を伴うため、出来不出来があります。私たちはこれらライブラリ作製とDNAショートリードのシークエンシングは外注しましたが、ゲノム上で数kbp離れた配列どうしを見つけるためのライブラリ(メートペアライブラリ、mate pair library)には問題も発生して、ゲノム配列ドラフトを作成するに必要な量のリードを得るには結局一年以上費やしました。そして、このように集めたリード情報をコンピュータ解析でつなぎ合わせて染色体レベルの配列を作製する作業は長田さんが実施しました(図2)。
遺伝子のexon-intron構造を推定するにはmRNAの配列情報が必要になります。そこで、ゲノム配列用のDNAシークエンシングに並行して、mRNAの網羅的配列決定であるRNA-seqも実施しました。RNA-seqは外注せずに黒田さんたちが自前でやってくれました(図2)。
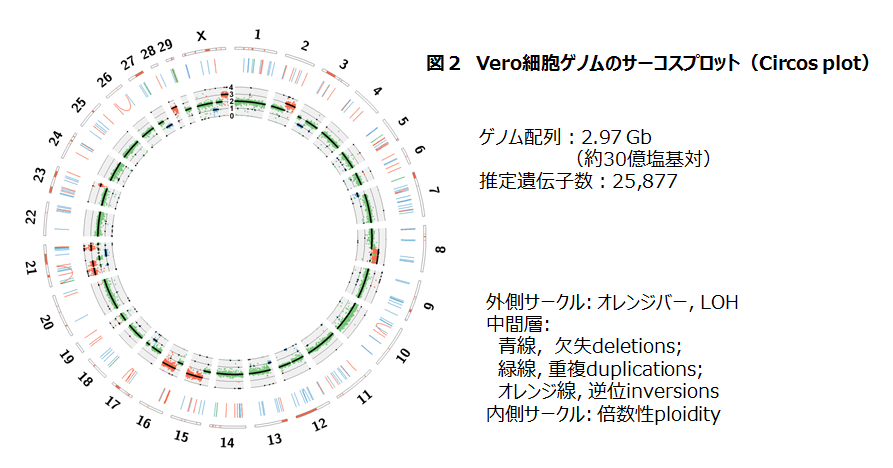
正常個体に比べると培養細胞のゲノム構造は不安定であり、Vero細胞においても核型解析レベルでも多くの染色体転座が観察されます。しかし、大規模並行型シーケンサ(massively parallel sequencer; いわゆる次世代型シーケンサnext generation sequencerの一つ)から得られる短いリードから再構築したVero細胞ゲノムの配列には、核型解析で見出された染色体転座は一つも現れてこないという問題に直面しました。
この問題に直面した当初こそ少々戸惑いましたが、このことは使用している技術を考えるとむしろ当然の結果だったと思われます。
現行の手法ですと、二倍体のゲノムの配列であっても典型的なものを一倍体のゲノムの配列として提示します(生物種を代表して世に提示されているゲノム配列はヒトを含めて有核生物全てでそうです)。典型的配列を得るために解析のフィルターを強くすると、相同染色体のうちの一本にしか起こっていない転座ははじかれます。といって、フィルターを甘くすると非典型配列がどんどん紛れ込んでアセンブル配列を作成できませんでした。さらに、染色体転座は反復配列が多い部分で起こりやすいことも転座部位を見出せなかった大きな原因と考えられます。短いリード情報をいくら数多く集めても長い反復配列を確実に決定することは困難だからです。このような技術的限界も考え、Vero細胞の染色体転座部位をゲノム配列レベルで決定することは諦めました(余談5)。
本研究で得られたDNA配列データは論文規定に従って公的データバンクを通じて公開しております(http://www.ncbi.nlm.nih.gov/sra/?term=DRA002256)。
当該論文上梓後にassembly fileも以下サイトで公開しています。
アノテーション付き情報もDDBJに掲示しています。
長田さんの以下personal archiveからでも得ることができます。
https://sites.google.com/site/nosada17/Home/vero-scaffolds?authuser=0
9.アフリカミドリザルのゲノム配列公開
Vero細胞のゲノム配列を決定しようと私たちがそのプロジェクトを開始した2012年初頭の時点では、Vero細胞が由来する動物であるアフリカミドリザルのゲノム配列も未決定でした。そのため、ゲノム配列を新規に決定するという方向性を取らざるを得ませんでした。ところが、ゲノム配列決定では世界的に有名な米国ワシントン大のグループがアフリカミドリザル属の複数の種(生物種の話題に関しては後述します)のゲノム配列を2013年の6月に米国国立バイオテクノロジーセンター(National Center for Biotechnology Information; NCBI)のデータベースに公開し、2014年には注釈annotationも付きました(注1)。
私たちは、ベロ細胞のゲノム配列を決定することによってアフリカミドリザルのゲノム配列決定の一番乗りにもなると当初は考えていたので、アフリカミドリザルのゲノム配列公開を知ったときは先を越されたとショックを受けたのですが、よくよく考えるとアフリカミドリザルの複数の種のゲノム配列と比較できる状況となったことはむしろ歓迎すべきことであり、実際にいろいろな恩恵をうけることになりました。
10.Vero細胞のゲノム配列解読でわかったこと
Vero細胞のゲノム配列を解読すると、この細胞の特性と密接に関わると思われるゲノム構造上の性質がいろいろとわかってきました[1]。以下にそれらを羅列的に紹介します。
(1)Vero細胞はAfrican green monkeysのChlorocebus sabaeusに由来する
Vero細胞が樹立された1960年代の時点では、アフリカミドリザルは一つの生物種Cercopithecus aethiopsとしてまとめられていました。しかし、分子生物学的解析手法の導入などに伴い、アフリカミドリザルの分類上の属はCercopithecusからChlorocebusに移され、さらに、複数の種からなっていると考えられるようになりました(よって、生物種を意識してアフリカミドリザル全体を表すとき、英語ではAfrican green monkeysと複数形でいう)[17, 18]。
では、Vero細胞は現在の分類法のどの動物種に由来しているのでしょうか?この問いに答えるにはミトコンドリアのゲノムの分子系統樹解析molecular phylogenetic analysisが適しています。旧世界ザル(old world monkeys;アジアおよびアフリカに棲む狭鼻下サル亜目Catarrhiniのサル)においては、核ゲノムに比べてミトコンドリアゲノムの変異率が各段に高いことが知られており、近接した生物種の間での遺伝学的統樹解析にはミトコンドリアゲノムのほうが高解像度の結果を与えるためです[18]。ミトコンドリアゲノム配列が参照できる4種のAfrican green monkeysとの比較解析により、Vero細胞は西アフリカのサバンナ地方を中心に棲息するChlorocebus sabaeusに由来していると私たちは結論しました(図3)。
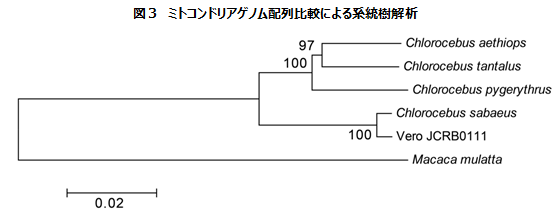
また、上述したように核型解析と性染色体のゲノム配列解析により、雌ザル由来であることも明確になったので、Vero細胞はメスのChlorocebus sabaeusに由来していることが樹立後約半世紀をへて明らかになったことになります。
(2)ウイルス抑制に働くI型インターフェロン遺伝子クラスターの欠失
細胞からウイルスを排除する仕組みにはさまざまなものがありますが、脊椎動物のどのタイプの細胞にでもほぼ普遍的に存在する仕組みの一つにI型インターフェロンの産生と応答があります。
ウイルスに感染した細胞はI型インターフェロンを生産し、それは感染細胞自身に直接働きかけて細胞内ウイルスを排除する作用と他の種類の細胞(特に免疫担当細胞)に働きかけて間接的に感染細胞を排除する作用を発揮します。動物個体のウイルス対応においてはどちらの作用も重要ですが、他の種類の細胞が同じ細胞皿中にないような培養細胞感染実験では感染細胞自身に直接働きかける作用だけということになります。
ウイルス感染した細胞から放出されたI型インターフェロンは、細胞膜上にあるI型インターフェロン受容体に結合すると多様な細胞内事象を引き起こします。その結果として、ウイルスゲノム情報を発現するために必須であるRNA合成やタンパク質合成が阻害され、ウイルス増殖が抑えられるようになります。
同一生物種に発現している機能的に似た働きを持ちアミノ酸配列的にもお互いに似たタンパク質をアイソフォームisoformと呼びます。I型インターフェロンには多くのアイソフォームがあり、それぞれに遺伝子があるのですが、これら遺伝子は染色体上の限られた位置にかたまって存在しています。このI型インターフェロン遺伝子クラスターは、ヒトでは9番染色体短腕領域(9p22)にあり、この領域はアフリカミドリザルでは12番染色体の領域に相当します。
ゲノム解析の結果、Vero細胞の12番染色体に900万塩基対(9 Mbp)に渡る大きな欠失、それも二本の相同染色体の両方で欠失があることが判明しました。Vero細胞のI型インターフェロン遺伝子のいくつかが失われていることは古典的なDNAハイブリダイゼーション法(サザンブロット法Southern blotting)によってすでにしめされていたので[19]、今回の全ゲノム配列決定は、過去の報告を確認したうえで、遺伝子クラスター全てがごっそりと欠失していることをゲノムDNA配列レベルで明確にしたことになります。さらに、以下に述べるような従来全く知られていなかったことも明らかになってきました。
(3)細胞周期のブレーキ役の欠失
細胞周期のブレーキ役として機能する二つのサイクリン依存性キナーゼ阻害因子をコードする遺伝子CDKN2A, CDKN2Bは、ヒトゲノムにおいてI型インターフェロン遺伝子クラスターの近傍に存在しており、この二つの遺伝子中の変異がいろいろなヒト癌細胞のゲノムで起こっていることが知られています[20, 21]。Vero細胞で欠失した9 Mbp領域のなかにはCDKN2A, CDKN2Bも含まれていることが判明しました。
動物個体を成す細胞は他の細胞と協調しつつ増殖・分化し、これ以上増える必要がない状況では細胞分裂を休止します。何かの原因で細胞周期制御メカニズムが破綻して細胞増殖がとめどなく進むようになると癌などの病気につながります。
細胞周期制御メカニズムが破綻すれば、それだけで無限に分裂増殖が可能な不死化細胞となるわけでもありませんし、不死化細胞が必ずしも造腫瘍性をもつ癌細胞というわけでもありません。変化としては、細胞周期制御機構の破綻 --> 細胞の不死化(例えば細胞分裂限度数を規定するテロメアの修復能の獲得は必須)--> 造腫瘍性、という順番で進むと思われます。これら一連の変化でゲノムの変異が表裏一体として起こっています(余談6)。
一次細胞培養に供されたアフリカミドリザル摘出腎臓細胞のごく一部にCDKN2A, CDKN2Bの欠失が起こって細胞周期制御が破綻し、さらなるゲノム変化が積み重なって不死化細胞株Vero細胞へとつながっていったと思われます。
イヌ腎臓由来MDCK細胞や新たに樹立したイヌ腎臓由来不死化細胞株でもCDKN2A, CDKN2Bに欠失があることが2015年に報告されています[22]。